
Biolinux
Biolinux è una distribuzione Linux basata su Ubuntu e il desktop Unity che rende il pc una workstation potente e flessibile adatta per l’uso nella bioinformatica. Per questo si presenta con più di 500 programmi bioinformatici richiamabili da un apposito menu semplificato, offre l’accesso a una ricca documentazione sull’argomento e contiene diversi dati campione per poter testare i programmi.
Versione 7
Questa versione si presenta con:
- Sistema basato su Ubuntu 12..04 LTS, la release con supporto lungo di Ubuntu
- L’ambiente popolare Galaxy è installato e già configurato per l’utilizzo
- Contiene il software PredictProtein della Rost Lab
- Comprende le versioni più recenti del software per l’attività microbica (QIIME, Mothur, AmpliconNoise, RDP-classifier, ChimeraSlayer)
- Include i software Velvet, Tophat, Cufflinks e Bowtie
- Continua il supporto a tool classici di analisi bioinformatica: EMBOSS, BLAST, BLAST+, Artemis, ARB, e MrBAYES.
- Offre un design modulare che permette meglio di integrarsi in ubuntu e sui sitemi cloud
- Si può aggiornare direttamente dalla versione 6 senza problemi di sorta
Risorse
La distribuzione può essere scaricata da (2,6GB): http://nebc.nerc.ac.uk/downloads/bio-linux-7.0.3.iso
Il sito web della distribuzione è: http://envgen.nox.ac.uk/tools/bio-linux
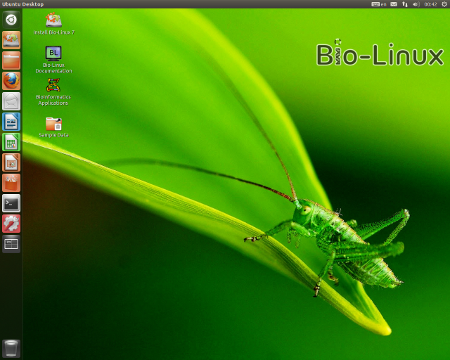
Screenshot
Conclusioni
Esistono molte distribuzioni Linux specializzate per offrire il meglio possibile per un determinato settore (es: networking, recovery, …), e BioLinux non è da meno nel suo campo.
Se siete ricercatori universitati o comunque lavorate nel settore della bioinformatica BioLinux è il coltellino svizzero del caso 😉




26 Novembre 2012 at 11:46
un ricercatore universitario che usa unity.. è un’idiozia solo a pensarlo
26 Novembre 2012 at 12:14
Al ricercatore universitario solitamente serve poter usare i programmi (a volte molto specifici e di nicchia) per la propria ricerca (in questo specifico campo ad esempio l’analisi microbica, la sequenziazione del DNA, ecc) per cui l’interfaccia per raggiungere questo scopo è solo una questione di abitudine (e fortunatamente in Linux si può scegliere tra molte interfaccie grafiche differenti nel caso in cui quella proposta di default non sia di gradimento).